决策树思想(基于性能评估思考)
以性能评估为目的,通过决策树评估出最重要的监控指标。
一、构建数据
1、样本数据形式
构建数据的工作主要是生成一条条的样本数据,现在就以执行时间为标记label,以下监控数据为特征来学习决策树。
| 序号 | 特征 | 名称 | 备注 |
|---|---|---|---|
| 1 | shared_replay_overhead | share memory冲突的次数 | share memory分配bank给线程组(半个wrap16个线程)时采用k路-组划分的原理,会导致冲突 |
| 2 | shared_load|store | 多核处理器上每个wrap执行Share Memory的load或者store指令的条数 | 每个wrap里的线程执行同样的指令集 |
| 3 | inst_replay_overhead | 指令重复计算的次数 | ?? |
| 4 | l1_global_load_hit | 访问GM时L1的命中次数 | L1作为全局load指令的cache,该项表示load GM时命中L1而不需要访问GM的次数 |
| 5 | l1_global_load_miss | 访问GM时L1的未命中次数 | 该项表示load GM时未命中L1的次数 |
| 6 | gld_request | 每个wrap里的线程执行GM的load的次数 | 未命中L1的线程需要访问GM,因此执行GM的load |
| 7 | gst_request | 每个wrap里的线程执行GM的store的次数 | 每个wrap里的多个线程执行相同的指令集 |
所有的样本数据都是“label,特征1,特征2,···”的形式,单位统一。
2、归一化操作
读取数据集,按特征值做0-1规划。因为很多执行时间、指标的波动性非常大,影响决策结果,故必须做归一化操作。
二、数据预分析
1、信息熵的计算
信息熵是用来表明当前信息的不确定性的,信息熵越小,确定性越高。如何计算信息熵?实际上就是label的期望值:假设有N个样本,M个label值,第i个label值出现的概率为p_i,那么当前的信息熵即
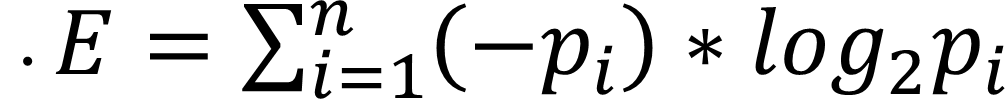
2、数据集的划分
根据特征划分数据集,当选择的特征划分出的数据集信息熵最小时,就是最佳特征了。
指定一个特征项F,做如下操作:
-
获取它所有可能的值(V1,V2,···,Vn)
-
针对每一项Vi做如下操作:
- 从原始数据集中过滤出所有特征项F值为Vi的数据,生成新的数据子集
- 计算新的数据子集的信息熵Ei
- 计算新的子集在原始数据集中所占比例Pi
-
当前特征项的信息熵为所有子集的信息熵的期望值
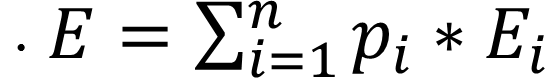
3、最大信息增益
由于信息熵越大就说明信息越不确定,所以我们需要获取最小的信息熵。因此,当前信息熵与某个特征值划分出的数据集的信息熵差值最大时,即所谓的最大信息增益。
针对每个特征值计算划分子集后的信息熵,得到最大信息增益的那个特征值。
double maxEntropyGain = 0.0;
int bestFeature;
for(int i : features){
if(baseEntropy-newEntropy > maxEntropyGain){
maxEntropyGain = baseEntropy-newEntropy;
bestFeature = i;
}
}
三、决策树
1、构建决策树
在预分析里,我们已经知道了如何找出最佳特征值,并且知道了如何根据特征值划分数据集。这个时候,一个数据集就被该特征值划分成了N个数据子集,N表示该特征值可能的取值。
至此,通过消耗一个特征值,我们将数据集划分成了一颗N叉树,根节点为包含所有特征的所有数据记录。接下来需要做的就是按照相同的方法递归处理每个子节点了。
最终停止的条件有两种情况:
- 当前节点中的数据全部具有相同的label值
- 特征值耗尽,但是当前节点中label值仍然有多个
第一种情况可以直接决定记录的类别。第二种情况则需要统计label值最多的那一类,或者算期望值。
2、过拟合
过拟合实际上就是过分划分导致严格按照特征的值进行分类,稍微出现值不同就会产生误判。具体表现为训练集上的样本匹配率非常高,而验证集上的匹配率却非常低。
数据样本小最容易导致过拟合,决策树中最简单高效的控制过拟合的方法有:
- 控制每个树节点的样本个数,小于阈值(经验值最少为100)时不再划分子集
- 控制树的高度
在划分数据集时判断即可。
附Java代码
package data;
import java.io.*;
import java.util.ArrayList;
import java.util.HashMap;
import java.util.HashSet;
/**
* 计算数据集的信息熵。需要在归一化后进行该操作。
* 迭代计算信息熵,当选取的特征值让信息熵最小即可终止计算
* Created by zx on 2017/2/20.
*/
class Record{
double label;
ArrayList<Double> features;
Record(double label, double[] initFeatures){
this.label = label;
this.features = new ArrayList<>();
for(int i = 0; i < initFeatures.length; i++)
features.add(initFeatures[i]);
}
public double getFeature(int index){
return features.get(index);
}
public double removeFeature(int index){
return features.remove(index);
}
}
class RegressionNode{
// 是否是叶子节点,1. 全部的label值相同;2. 没有特征值了
boolean isLeaf = false;
// 是否全部的label值相同
boolean isSameLabel = false;
//该节点包含的record数目
int recordNum = 0;
//用于划分的特征值在当前特征值组中的序列
int splitFeatureIndex = -1;
//子节点的个数
int childNum = 0;
//当前特征值的个数
int featuresNum = 0;
//用于划分的特征值的具体数值,与每个child对应
ArrayList<Double> feature2Child = new ArrayList<>();
//子节点
ArrayList<RegressionNode> children = new ArrayList<>();
// 如果全部的label值不同,则保留在此
ArrayList<Record> remainRecords = new ArrayList<>();
public String toString(){
StringBuilder stringBuilder = new StringBuilder();
stringBuilder.append("Node with ");
stringBuilder.append(recordNum + " records. ");
stringBuilder.append(childNum + " children. ");
stringBuilder.append(featuresNum + " features. ");
if(isSameLabel){
stringBuilder.append("All same labels. ");
}else{
stringBuilder.append("Not same labels. ");
}
if(splitFeatureIndex != -1){
stringBuilder.append("Best feature is " + splitFeatureIndex);
}
return stringBuilder.toString();
}
public void print(){
System.out.println(this);
for(int i = 0; i < children.size(); i++){
children.get(i).print();
}
}
}
public class RegressionTree {
/***** 重要参数 *****/
// 每个节点的最少样本数
int NUM_SAMPLES_IN_NODE = 100;
// 树的最大高度
int MAX_HEIGHT = 5;
/*** ***/
ArrayList<Record> totalRecords = new ArrayList<>();
int _NFeatures = 0;
RegressionNode root = new RegressionNode();
/**
* 全部的数据要放置在内存,这个很耗时啊···
* @param input
* @return
*/
public void startRegression(String input, int numFeatures) {
readData(input);
_NFeatures = numFeatures;
regression(totalRecords, _NFeatures, root);
root.print();
}
public void regression(ArrayList<Record> records, int numFeatures, RegressionNode node){
node.recordNum = records.size();
node.featuresNum = numFeatures;
//防止过拟合
if(records.size() < NUM_SAMPLES_IN_NODE || _NFeatures-numFeatures > MAX_HEIGHT){
node.isLeaf = true;
node.remainRecords = records;
node.isSameLabel = isSameLabels(records);
return;
}
//特征值个数为0了
if(numFeatures == 0) {
node.isLeaf = true;
node.remainRecords = records;
return;
}
//包含的record所有label都相同了
if(isSameLabels(records)){
node.isLeaf = true;
node.isSameLabel = true;
return;
}
double baseEntropy = computeEntropy(records);
double maxEntropyGain = 0;
int bestFeature = 0;
for(int i = 0; i < numFeatures; i++){
double newEntropy = computeFeatureEntropy(i, records);
if(baseEntropy-newEntropy > maxEntropyGain){
maxEntropyGain = baseEntropy - newEntropy;
bestFeature = i;
}
}
node.splitFeatureIndex = bestFeature;
HashMap<Double, ArrayList<Record>> feature2SubRecords = new HashMap<>();
for(int i = 0; i < records.size(); i++){
Record record = records.get(i);
double key = record.removeFeature(bestFeature);
if(feature2SubRecords.containsKey(key)){
ArrayList<Record> value = feature2SubRecords.get(key);
value.add(record);
feature2SubRecords.put(key, value);
}else{
ArrayList<Record> value = new ArrayList<>();
value.add(record);
feature2SubRecords.put(key, value);
}
}
node.childNum = feature2SubRecords.size();
for(double key: feature2SubRecords.keySet()) {
RegressionNode childNode = new RegressionNode();
node.children.add(childNode);
node.feature2Child.add(key);
ArrayList<Record> values = feature2SubRecords.get(key);
regression(values, numFeatures - 1, childNode);
}
}
public boolean isSameLabels(ArrayList<Record> records){
for(int i = 0; i < records.size()-1; i++){
if(records.get(i).label != records.get(i+1).label)
return false;
}
return true;
}
public double computeEntropy(ArrayList<Record> entroyRecords){
double entropy = 0.0;
HashMap<Double, Integer> labelNums = new HashMap<>();
for (int i = 0; i < entroyRecords.size(); i++) {
Record record = entroyRecords.get(i);
if (labelNums.containsKey(record.label)) {
int old = labelNums.get(record.label);
labelNums.put(record.label, old + 1);
} else {
labelNums.put(record.label, 1);
}
}
for (Double key : labelNums.keySet()) {
double p = labelNums.get(key) / (double) labelNums.size();
entropy -= (p * Math.log(p) / Math.log(2));
}
return entropy;
}
public void readData(String input) {
try {
File inputFile = new File(input);
FileReader inputReader = new FileReader(inputFile);
BufferedReader bufferedReader = new BufferedReader(inputReader);
String s;
while ((s = bufferedReader.readLine()) != null) {
String[] parts = s.split(",");
double label = Double.parseDouble(parts[0]);
double[] features = new double[parts.length - 1];
for (int i = 1; i < parts.length; i++)
features[i - 1] = Double.parseDouble(parts[i]);
Record record = new Record(label, features);
totalRecords.add(record);
}
} catch (FileNotFoundException e) {
System.out.println("Input File Not Found. " + e.getMessage());
} catch (IOException e) {
System.out.println("Read File Error. " + e.getMessage());
}
}
public double computeFeatureEntropy(int chooseIndex, ArrayList<Record> records){
if(chooseIndex < 0 || chooseIndex >= _NFeatures){
System.out.println("Choose Features Error.");
return 0;
}else{
double entropy = 0.0;
HashSet<Double> featurePossibleValues = new HashSet<>();
//获取该特征所有可能的值
for(int i = 0; i < records.size(); i++){
featurePossibleValues.add(records.get(i).getFeature(chooseIndex));
}
//对每个可能的值,过滤出所有特征项值与之相等的记录,计算新的样本集的信息熵
for(double value: featurePossibleValues){
ArrayList<Record> entropyRecords = new ArrayList<>();
for(int i = 0; i < records.size(); i++){
Record record = records.get(i);
if(record.features.get(chooseIndex) == value){
entropyRecords.add(record);
}
}
//获取该子集的信息熵,乘以该子集数据量所占比例,得到整个特征值的信息熵
double thisEntropy = computeEntropy(entropyRecords);
entropy += thisEntropy * (entropyRecords.size() / (double)records.size());
}
return entropy;
}
}
}

